5 Pochodne drzew decyzyjnych
Przykład zastosowania drzew decyzyjnych na zbiorze iris w poprzednich przykładach może skłaniać do przypuszczenia, że drzewa decyzyjne zawsze dobrze radzą sobie z predykcją wartości wynikowej. Niestety w przykładach nieco bardziej skomplikowanych, gdzie chociażby klasy zmiennej wynikowej nie są tak wyraźnie separowalne, drzewa decyzyjne wypadają gorzej w porównaniu z innymi modelami nadzorowanego uczenia maszynowego.
I tak u podstaw metod bazujących na prostych drzewach decyzyjnych stał pomysł, że skoro jedno drzewo nie ma wystarczających własności predykcyjnych, to może zastosowanie wielu drzew połączonych w pewien sposób poprawi je. Tak powstały metody bagging, random forest i boosting17. Należy zaznaczyć, że metody znajdują swoje zastosowanie również w innych modelach nadzorowanego uczenia maszynowego.
5.1 Bagging
Technika ta została wprowadzona przez Breiman (1996) i ma na celu zmniejszenie wariancji modelu pojedynczego drzewa. Podobnie jak technika bootstrap, w której statystyki są wyliczane na wielu próbach pobranych z tego samego rozkładu (próby), w metodzie bagging losuje się wiele prób ze zbioru uczącego (najczęściej poprzez wielokrotne losowanie próby o rozmiarze zbioru uczącego ze zwracaniem), a następnie dla każdej próby bootstrapowej buduje się drzewo. W ten sposób otrzymujemy \(B\) drzew decyzyjnych \(\hat{f}^1(x), \hat{f}^2(x),\ldots, \hat{f}^B(x)\). Na koniec poprzez uśrednienie otrzymujemy model charakteryzujący się większą precyzją \[\begin{equation} \hat{f}_{bag}(x)=\frac1B\sum_{b=1}^B\hat{f}^b(x). \end{equation}\]
Ponieważ podczas budowy drzew na podstawie prób bootstrapowych nie kontrolujemy złożoności, to w rezultacie każde z drzew może charakteryzować się dużą wariancją. Poprzez uśrednianie wyników pojedynczych drzew otrzymujemy mniejsze obciążenie ale również przy dostatecznie dużej liczbie prób (\(B\) często liczy się w setkach, czy tysiącach) zmniejszamy wariancję “średniej” predykcji z drzew. Oczywiście metodę tą trzeba dostosować do zadań klasyfikacyjnych, ponieważ nie istnieje średnia klasyfikacji z wielu drzew. W miejsce średniej stosuje się modę, czyli wartość dominującą.
Przyjrzyjmy się jak maszyna losuje obserwacje ze zwracaniem
n <- NULL
m <- NULL
for(i in 1:1000){
x <- sample(1:500, size = 500, replace = T)
y <- setdiff(1:500, x)
z <- unique(x)
n[i] <- length(z)
m[i] <- length(y)
}
mean(n)/500*100## [1] 63.2802## [1] 36.7198Faktycznie uczenie modelu metodą bagging odbywa się średnio na 2/3 obserwacji zbioru uczącego wylosowanych do prób bootstrapowych, a pozostała 1/3 (ang. out-of-bag) jest wykorzystana do oceny jakości predykcji.
Niewątpliwą zaletą drzew decyzyjnych była ich łatwa interpretacja. W przypadku metody bagging jest ona znacznie utrudniona, ponieważ jej wynik składa się z agregacji wielu drzew. Można natomiast ocenić ważność predyktorów (ang. variable importance). I tak, przez obserwację spadku \(RSS\) dla baggingu regresyjnego przy zastosowaniu danego predyktora w podziałach drzewa i uśrednieniu wyniku otrzymamy wskaźnik ważności predyktora dużo lepszy niż dla pojedynczego drzewa. W przypadku baggingu klasyfikacyjnego w miejsce \(RSS\) stosujemy indeks Gini’ego.
Implementacja R-owa metody bagging znajduje się w pakiecie ipred, a funkcja do budowy modelu nazywa się bagging (Peters and Hothorn 2018). Można również stosować funkcję randomForest pakietu randomForest (Liaw and Wiener 2002) - powody takiego działania wyjaśnią się w podrozdziale Lasy losowe.
Przykład 5.1 Tym razem cel zadania jest regresyjny i polega na ustaleniu miary tendencji centralnej ceny mieszkań w Bostonie na podstawie zmiennych umieszczonych w zbiorze Boston pakietu MASS (Venables and Ripley 2002). Zmienną zależną będzie mediana cen mieszkań na przedmieściach Bostonu (medv).
## crim zn indus chas nox rm age dis rad tax ptratio black lstat
## 1 0.00632 18 2.31 0 0.538 6.575 65.2 4.0900 1 296 15.3 396.90 4.98
## 2 0.02731 0 7.07 0 0.469 6.421 78.9 4.9671 2 242 17.8 396.90 9.14
## 3 0.02729 0 7.07 0 0.469 7.185 61.1 4.9671 2 242 17.8 392.83 4.03
## 4 0.03237 0 2.18 0 0.458 6.998 45.8 6.0622 3 222 18.7 394.63 2.94
## 5 0.06905 0 2.18 0 0.458 7.147 54.2 6.0622 3 222 18.7 396.90 5.33
## 6 0.02985 0 2.18 0 0.458 6.430 58.7 6.0622 3 222 18.7 394.12 5.21
## medv
## 1 24.0
## 2 21.6
## 3 34.7
## 4 33.4
## 5 36.2
## 6 28.7set.seed(2019)
boston.train <- Boston %>%
sample_frac(size = 2/3)
boston.test <- setdiff(Boston, boston.train)Aby móc porównać wyniki predykcji z metody bagging, najpierw zostanie zbudowane jedno drzewo decyzyjne w oparciu o algorytm CART.
library(rpart)
library(rpart.plot)
boston.rpart <- rpart(medv~., data = boston.train)
x <- summary(boston.rpart)## Call:
## rpart(formula = medv ~ ., data = boston.train)
## n= 337
##
## CP nsplit rel error xerror xstd
## 1 0.49839799 0 1.0000000 1.0086928 0.10259521
## 2 0.15725128 1 0.5016020 0.5442932 0.06125724
## 3 0.07485605 2 0.3443507 0.4031978 0.05139310
## 4 0.03672387 3 0.2694947 0.3127794 0.04599170
## 5 0.03552748 4 0.2327708 0.2974517 0.04560807
## 6 0.01695185 5 0.1972433 0.2553208 0.04022970
## 7 0.01422576 6 0.1802915 0.2713816 0.04099092
## 8 0.01103490 7 0.1660657 0.2744789 0.04107777
## 9 0.01000000 8 0.1550308 0.2720415 0.04119266
##
## Variable importance
## rm lstat indus ptratio crim age nox dis zn tax
## 33 19 9 8 7 6 6 5 3 2
## rad chas
## 1 1
##
## Node number 1: 337 observations, complexity param=0.498398
## mean=22.69792, MSE=79.32964
## left son=2 (286 obs) right son=3 (51 obs)
## Primary splits:
## rm < 6.92 to the left, improve=0.4983980, (0 missing)
## lstat < 9.725 to the right, improve=0.4424796, (0 missing)
## indus < 6.66 to the right, improve=0.2796065, (0 missing)
## ptratio < 19.65 to the right, improve=0.2600149, (0 missing)
## nox < 0.6695 to the right, improve=0.2346383, (0 missing)
## Surrogate splits:
## ptratio < 14.55 to the right, agree=0.884, adj=0.235, (0 split)
## lstat < 4.915 to the right, agree=0.878, adj=0.196, (0 split)
## zn < 87.5 to the left, agree=0.864, adj=0.098, (0 split)
## indus < 1.605 to the right, agree=0.864, adj=0.098, (0 split)
## crim < 0.013355 to the right, agree=0.852, adj=0.020, (0 split)
##
## Node number 2: 286 observations, complexity param=0.1572513
## mean=20.04266, MSE=37.17489
## left son=4 (114 obs) right son=5 (172 obs)
## Primary splits:
## lstat < 14.405 to the right, improve=0.3954065, (0 missing)
## nox < 0.6695 to the right, improve=0.3012249, (0 missing)
## crim < 8.37969 to the right, improve=0.2817286, (0 missing)
## ptratio < 20.15 to the right, improve=0.2392532, (0 missing)
## dis < 2.4737 to the left, improve=0.2295258, (0 missing)
## Surrogate splits:
## age < 84.3 to the right, agree=0.808, adj=0.518, (0 split)
## dis < 2.23935 to the left, agree=0.773, adj=0.430, (0 split)
## crim < 4.067905 to the right, agree=0.762, adj=0.404, (0 split)
## nox < 0.5765 to the right, agree=0.762, adj=0.404, (0 split)
## indus < 16.57 to the right, agree=0.759, adj=0.395, (0 split)
##
## Node number 3: 51 observations, complexity param=0.07485605
## mean=37.58824, MSE=54.4677
## left son=6 (34 obs) right son=7 (17 obs)
## Primary splits:
## rm < 7.47 to the left, improve=0.72041550, (0 missing)
## lstat < 3.99 to the right, improve=0.34223650, (0 missing)
## ptratio < 15.05 to the right, improve=0.21227430, (0 missing)
## rad < 2.5 to the left, improve=0.10053340, (0 missing)
## tax < 267 to the right, improve=0.07935891, (0 missing)
## Surrogate splits:
## lstat < 3.99 to the right, agree=0.824, adj=0.471, (0 split)
## indus < 1.215 to the right, agree=0.706, adj=0.118, (0 split)
## chas < 0.5 to the left, agree=0.706, adj=0.118, (0 split)
## tax < 225 to the right, agree=0.706, adj=0.118, (0 split)
## crim < 1.3713 to the left, agree=0.686, adj=0.059, (0 split)
##
## Node number 4: 114 observations, complexity param=0.03552748
## mean=15.33333, MSE=21.50994
## left son=8 (77 obs) right son=9 (37 obs)
## Primary splits:
## crim < 0.69916 to the right, improve=0.3873341, (0 missing)
## nox < 0.6615 to the right, improve=0.3541892, (0 missing)
## dis < 2.3497 to the left, improve=0.3182514, (0 missing)
## ptratio < 19.45 to the right, improve=0.3102781, (0 missing)
## tax < 567.5 to the right, improve=0.2823826, (0 missing)
## Surrogate splits:
## ptratio < 19.95 to the right, agree=0.895, adj=0.676, (0 split)
## indus < 14.345 to the right, agree=0.868, adj=0.595, (0 split)
## nox < 0.5825 to the right, agree=0.868, adj=0.595, (0 split)
## tax < 397 to the right, agree=0.868, adj=0.595, (0 split)
## rad < 16 to the right, agree=0.860, adj=0.568, (0 split)
##
## Node number 5: 172 observations, complexity param=0.03672387
## mean=23.16395, MSE=23.11579
## left son=10 (82 obs) right son=11 (90 obs)
## Primary splits:
## lstat < 9.645 to the right, improve=0.24693150, (0 missing)
## rm < 6.543 to the left, improve=0.17749260, (0 missing)
## ptratio < 17.85 to the right, improve=0.07815189, (0 missing)
## nox < 0.5125 to the right, improve=0.07760816, (0 missing)
## tax < 267.5 to the right, improve=0.07238020, (0 missing)
## Surrogate splits:
## nox < 0.5125 to the right, agree=0.756, adj=0.488, (0 split)
## indus < 7.625 to the right, agree=0.750, adj=0.476, (0 split)
## rm < 6.26 to the left, agree=0.738, adj=0.451, (0 split)
## age < 65.25 to the right, agree=0.727, adj=0.427, (0 split)
## dis < 3.8824 to the left, agree=0.709, adj=0.390, (0 split)
##
## Node number 6: 34 observations
## mean=33.15882, MSE=13.41419
##
## Node number 7: 17 observations
## mean=46.44706, MSE=18.85661
##
## Node number 8: 77 observations, complexity param=0.0110349
## mean=13.33247, MSE=15.64998
## left son=16 (37 obs) right son=17 (40 obs)
## Primary splits:
## lstat < 20.1 to the right, improve=0.24481010, (0 missing)
## crim < 15.718 to the right, improve=0.23250740, (0 missing)
## dis < 2.0037 to the left, improve=0.17113480, (0 missing)
## nox < 0.6615 to the right, improve=0.11757680, (0 missing)
## rm < 5.5675 to the left, improve=0.09054612, (0 missing)
## Surrogate splits:
## dis < 1.9733 to the left, agree=0.792, adj=0.568, (0 split)
## rm < 5.632 to the left, agree=0.727, adj=0.432, (0 split)
## age < 95.35 to the right, agree=0.675, adj=0.324, (0 split)
## crim < 9.08499 to the right, agree=0.662, adj=0.297, (0 split)
## black < 396.295 to the right, agree=0.623, adj=0.216, (0 split)
##
## Node number 9: 37 observations
## mean=19.4973, MSE=8.034858
##
## Node number 10: 82 observations
## mean=20.66098, MSE=6.55677
##
## Node number 11: 90 observations, complexity param=0.01695185
## mean=25.44444, MSE=27.29425
## left son=22 (83 obs) right son=23 (7 obs)
## Primary splits:
## age < 86.7 to the left, improve=0.1844883, (0 missing)
## lstat < 4.46 to the right, improve=0.1773076, (0 missing)
## dis < 3.0037 to the right, improve=0.1652768, (0 missing)
## crim < 0.628575 to the left, improve=0.1203635, (0 missing)
## nox < 0.5585 to the left, improve=0.1122403, (0 missing)
## Surrogate splits:
## nox < 0.5585 to the left, agree=0.978, adj=0.714, (0 split)
## dis < 2.1491 to the right, agree=0.978, adj=0.714, (0 split)
## crim < 0.643205 to the left, agree=0.967, adj=0.571, (0 split)
## indus < 16.57 to the left, agree=0.956, adj=0.429, (0 split)
## ptratio < 14.75 to the right, agree=0.956, adj=0.429, (0 split)
##
## Node number 16: 37 observations
## mean=11.2973, MSE=10.14026
##
## Node number 17: 40 observations
## mean=15.215, MSE=13.37128
##
## Node number 22: 83 observations, complexity param=0.01422576
## mean=24.79277, MSE=13.56694
## left son=44 (55 obs) right son=45 (28 obs)
## Primary splits:
## rm < 6.543 to the left, improve=0.3377388, (0 missing)
## lstat < 5.41 to the right, improve=0.2548210, (0 missing)
## tax < 267.5 to the right, improve=0.2210129, (0 missing)
## ptratio < 18.05 to the right, improve=0.1394682, (0 missing)
## dis < 6.4889 to the right, improve=0.1125739, (0 missing)
## Surrogate splits:
## lstat < 5.055 to the right, agree=0.783, adj=0.357, (0 split)
## ptratio < 15.75 to the right, agree=0.723, adj=0.179, (0 split)
## crim < 0.39646 to the left, agree=0.699, adj=0.107, (0 split)
## chas < 0.5 to the left, agree=0.687, adj=0.071, (0 split)
## age < 74.15 to the left, agree=0.687, adj=0.071, (0 split)
##
## Node number 23: 7 observations
## mean=33.17143, MSE=125.3192
##
## Node number 44: 55 observations
## mean=23.26545, MSE=8.880443
##
## Node number 45: 28 observations
## mean=27.79286, MSE=9.189949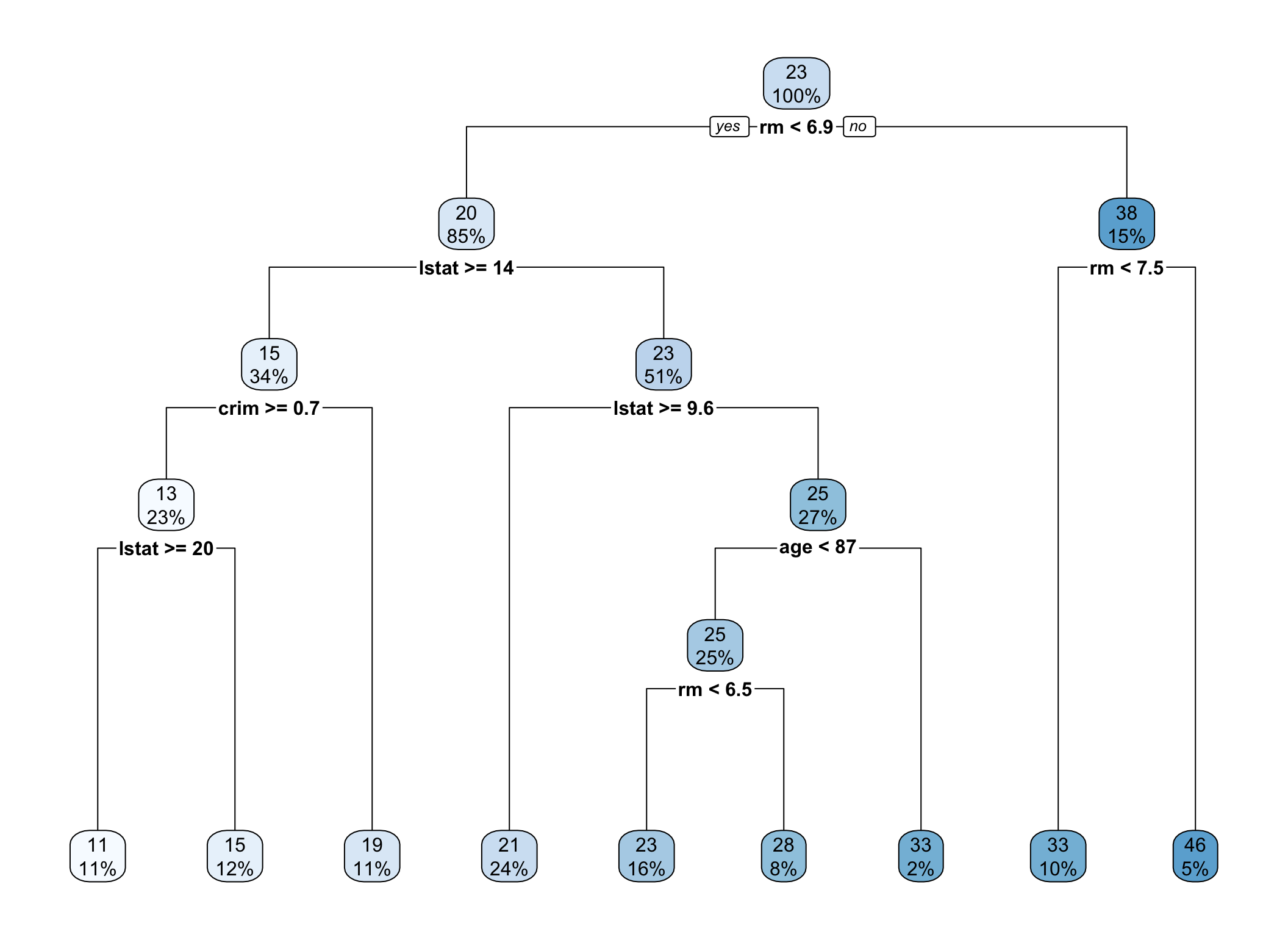
Rysunek 5.1: Drzewo regresyjne pełne
Przycinamy drzewo…
##
## Regression tree:
## rpart(formula = medv ~ ., data = boston.train)
##
## Variables actually used in tree construction:
## [1] age crim lstat rm
##
## Root node error: 26734/337 = 79.33
##
## n= 337
##
## CP nsplit rel error xerror xstd
## 1 0.498398 0 1.00000 1.00869 0.102595
## 2 0.157251 1 0.50160 0.54429 0.061257
## 3 0.074856 2 0.34435 0.40320 0.051393
## 4 0.036724 3 0.26949 0.31278 0.045992
## 5 0.035527 4 0.23277 0.29745 0.045608
## 6 0.016952 5 0.19724 0.25532 0.040230
## 7 0.014226 6 0.18029 0.27138 0.040991
## 8 0.011035 7 0.16607 0.27448 0.041078
## 9 0.010000 8 0.15503 0.27204 0.041193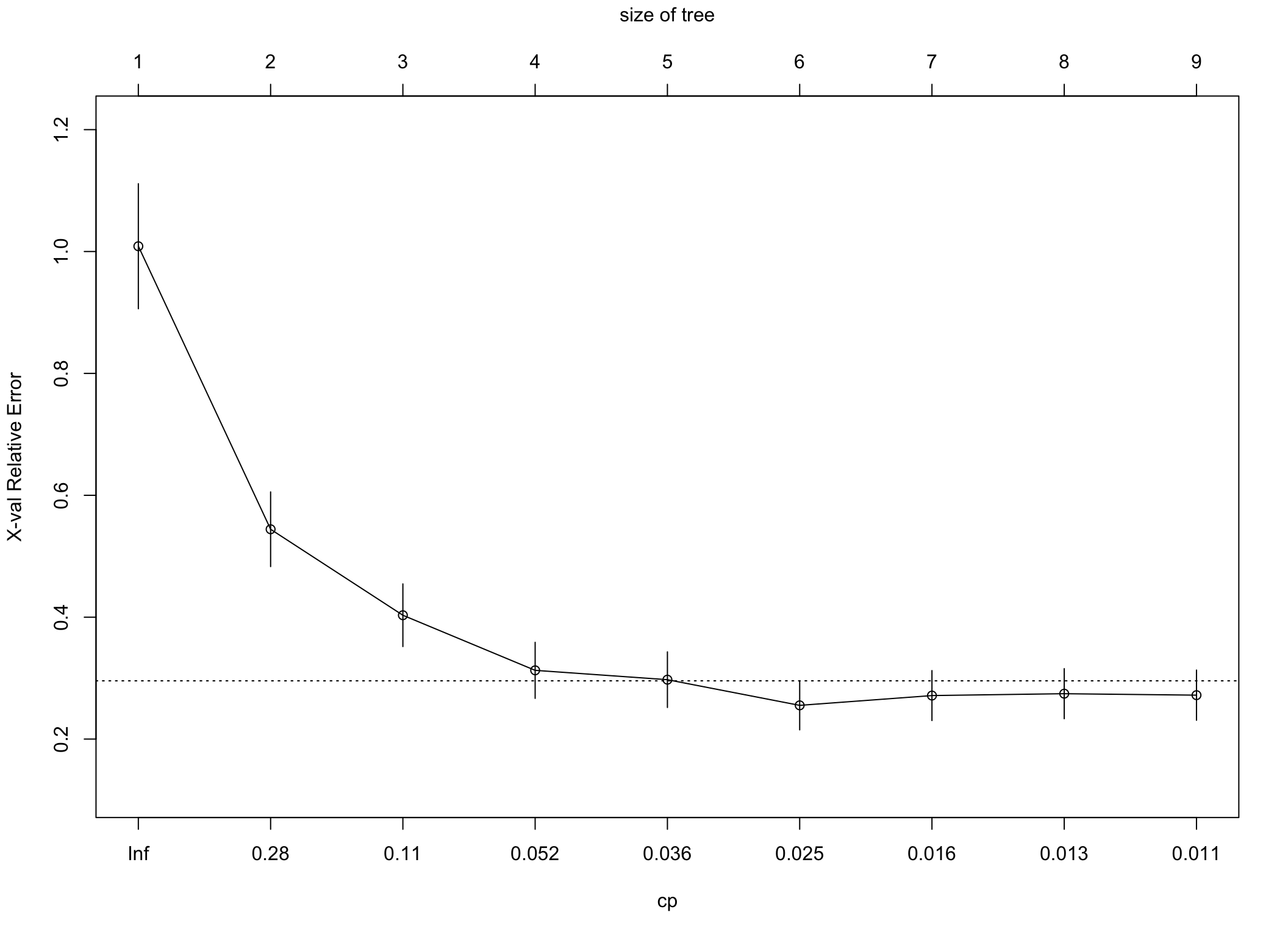
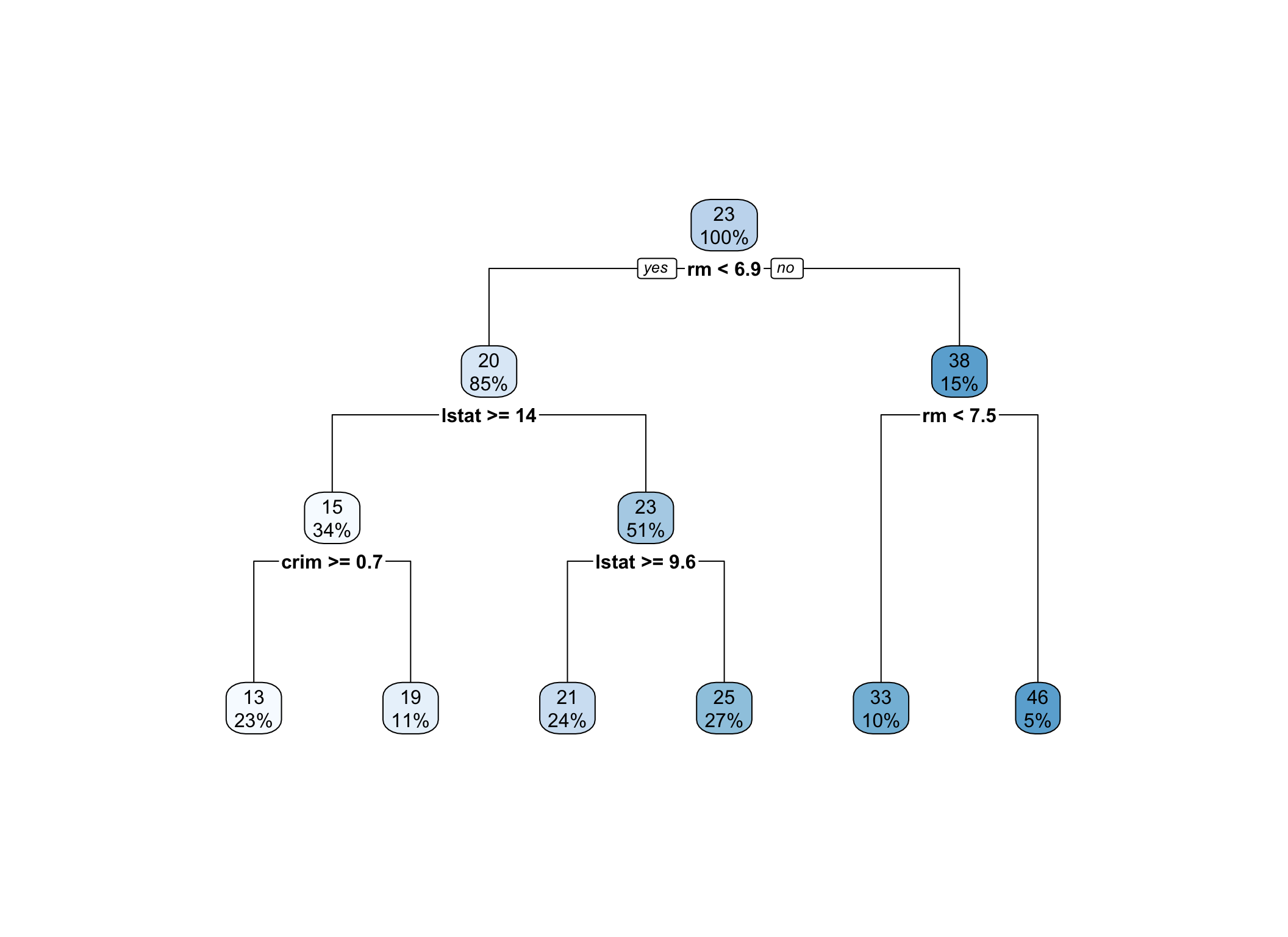
Rysunek 5.2: Drzewo regresyjne przycięte
Predykcja na podstawie drzewa na zbiorze testowym.
boston.pred <- predict(boston.rpart2, newdata = boston.test)
rmse <- function(pred, obs) sqrt(1/length(pred)*sum((pred-obs)^2))
rmse(boston.pred, boston.test$medv)## [1] 5.830722Teraz zbudujemy model metodą bagging.
library(randomForest)
boston.bag <- randomForest(medv~., data = boston.train,
mtry = ncol(boston.train)-1)
boston.bag##
## Call:
## randomForest(formula = medv ~ ., data = boston.train, mtry = ncol(boston.train) - 1)
## Type of random forest: regression
## Number of trees: 500
## No. of variables tried at each split: 13
##
## Mean of squared residuals: 12.03374
## % Var explained: 84.83Predykcja na podstawie modelu
## [1] 4.359119Zatem predykcja na podstawie modelu bagging jest nico lepsza niż z pojedynczego drzewa. Dodatkowo możemy ocenić ważność zmiennych użytych w budowie drzew.
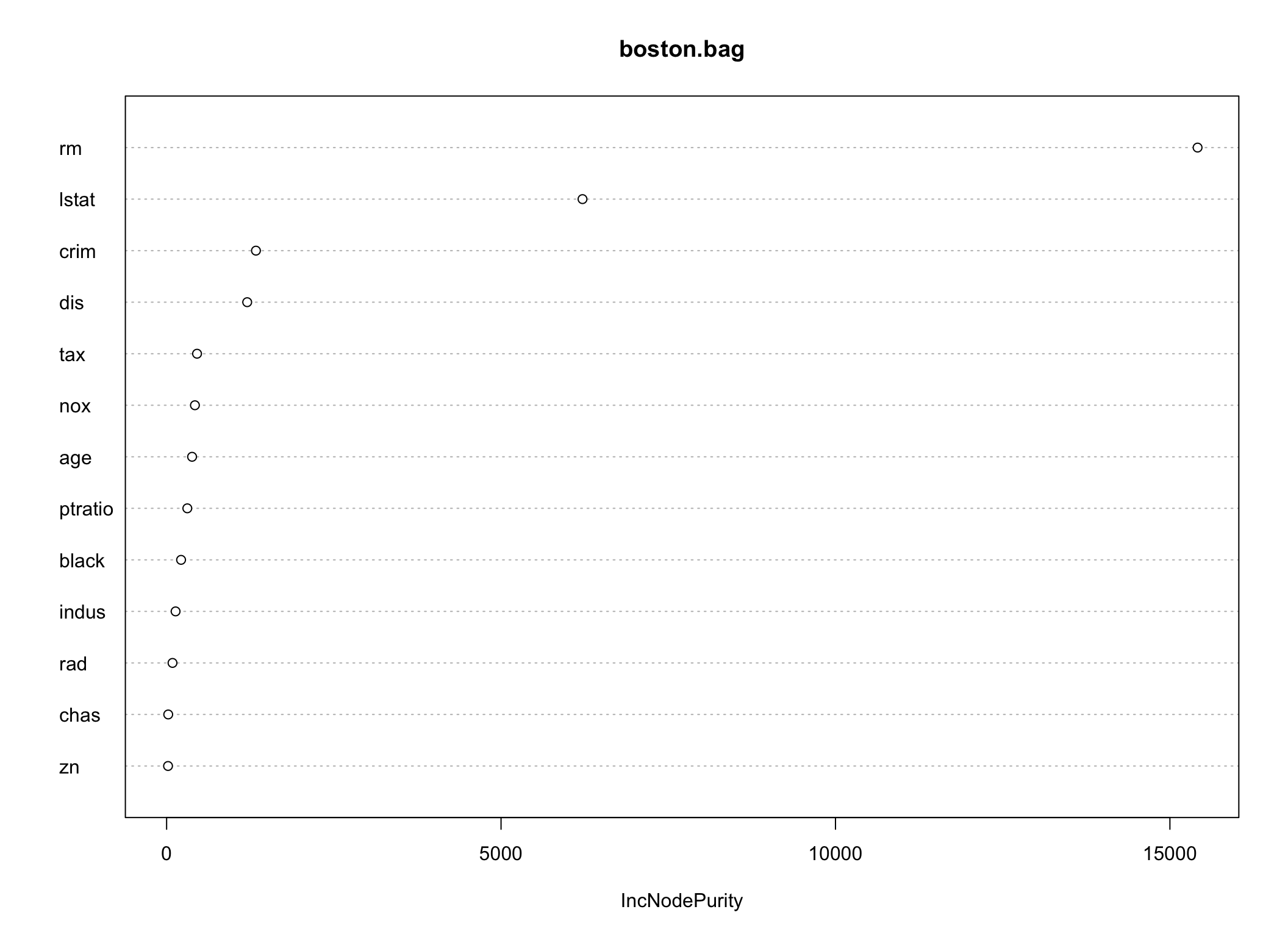
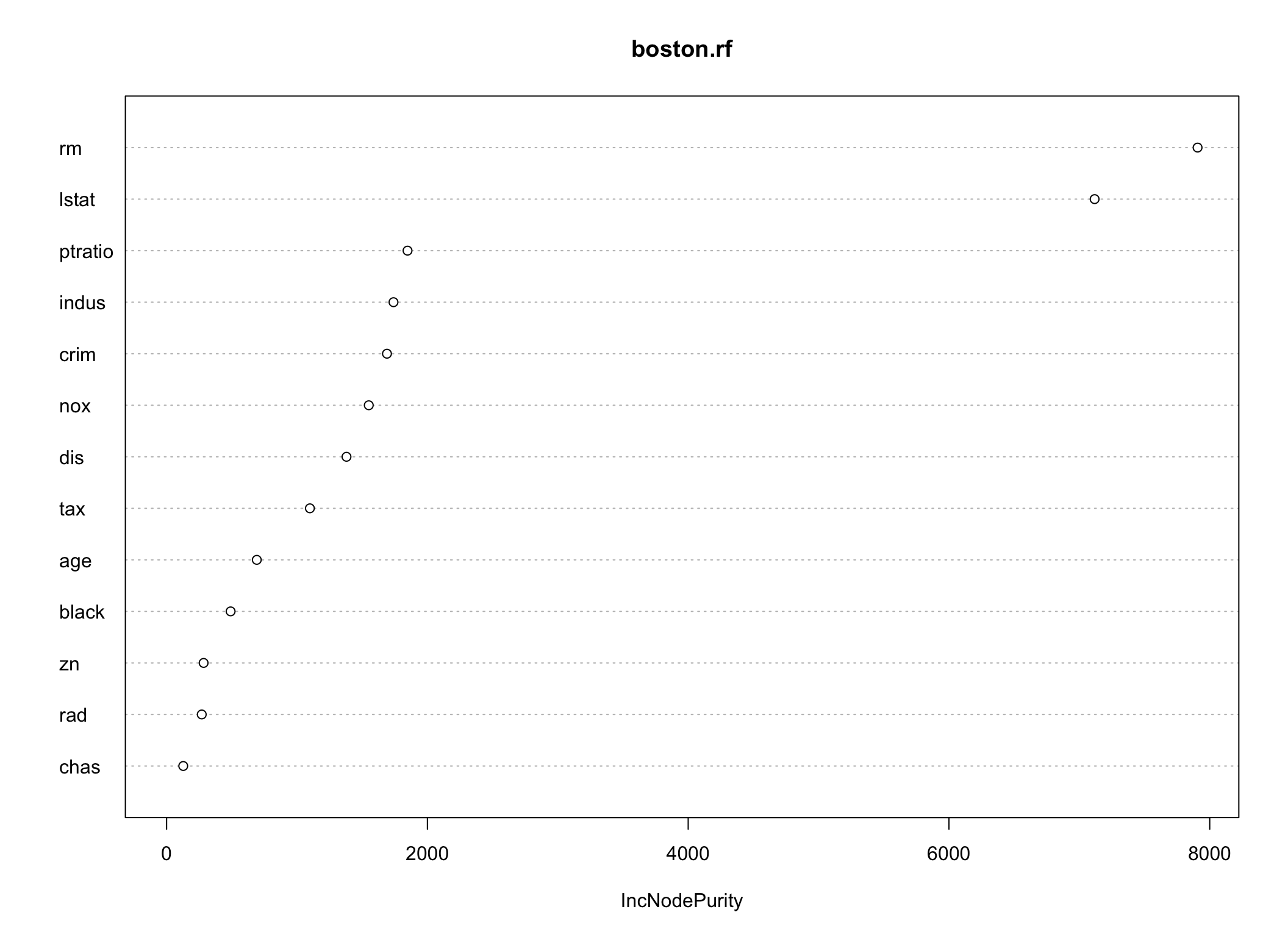
Rysunek 5.3: Wykres ważności predyktorów
## IncNodePurity
## crim 1335.62584
## zn 21.35274
## indus 134.28748
## chas 24.07230
## nox 423.26229
## rm 15413.69291
## age 380.78172
## dis 1204.86690
## rad 88.28151
## tax 454.99800
## ptratio 309.58412
## black 216.15512
## lstat 6217.95834## rm lstat indus ptratio crim age
## 16276.30598 9170.91941 4427.10554 4039.00112 3412.53062 3170.82658
## nox dis zn tax rad chas
## 3063.70694 2681.24858 1306.29569 800.17910 539.07271 262.60146
## black
## 63.78554W porównaniu do ważności zmiennych dla pojedynczego drzewa widać pewne różnice.
5.2 Lasy losowe
Lasy losowe są uogólnieniem metody bagging, polegającą na losowaniu dla każdego drzewa wchodzącego w skład lasu \(m\) predyktorów spośród \(p\) dostępnych, a następnie budowaniu drzew z wykorzystaniem tylko tych predyktorów (Ho 1995). Dzięki temu za każdy razem drzewo jest budowane w oparciu o nowy zestaw cech (najczęściej przyjmujemy \(m=\sqrt{p}\)). W przypadku modeli bagging za każdym razem najsilniejszy predyktor wchodził w skład zbioru uczącego, a co za tym idzie również uczestniczył w tworzeniu reguł podziału. Wówczas wiele drzew zawierało reguły stosujące dany atrybut, a wtedy predykcje otrzymywane za pomocą drzew były skorelowane. Dlatego nawet duża liczba prób bootstrapowych nie zapewniała poprawy precyzji. Implementacja tej metody znajduje się w pakiecie randomForest.
Przykład 5.2 Kontynuując poprzedni przykład 5.1 możemy zbudować las losowy aby przekonać się czy nastąpi poprawa predykcji zmiennej wynikowej.
##
## Call:
## randomForest(formula = medv ~ ., data = boston.train)
## Type of random forest: regression
## Number of trees: 500
## No. of variables tried at each split: 4
##
## Mean of squared residuals: 12.05123
## % Var explained: 84.81Porównanie MSE na próbach uczących pomiędzy lasem losowym i modelem bagging wypada nieco na korzyść bagging.
## [1] 3.79973Ważność zmiennych również się nieco różni.
5.3 Boosting
Rozważania na temat metody boosting zaczęły się od pytań postawionych w publikacji Kearns and Valiant (1989), czy da się na podstawie na podstawie zbioru słabych modeli stworzyć jeden dobry? Odpowiedzi pozytywnej na nie udzielili, najpierw Schapire (1990), a potem Breiman (1998). W metodzie boosting nie stosuje się prób bootstrapowych ale odpowiednio modyfikuje się drzewo wyjściowe w kolejnych krokach na tym samym zbiorze uczącym. Algorytm dla drzewa regresyjnego jest następujący:
- Ustal \(\hat{f}(x)=0\) i \(r_i=y_i\) dla każdego \(i\) w zbiorze uczącym.
- Dla \(b=1,2,\ldots, B\) powtarzaj:
- naucz drzewo \(\hat{f}^b\) o \(d\) regułach podziału (czyli \(d+1\) liściach) na zbiorze \((X_i, r_i)\),
- zaktualizuj drzewo do nowej “skurczonej” wersji \[\begin{equation} \hat{f}(x)\leftarrow \hat{f}(x)+\lambda\hat{f}^b(x), \end{equation}\]
- zaktualizuj reszty \[\begin{equation} r_i\leftarrow r_i-\lambda\hat{f}^b(x_i). \end{equation}\]
- Wyznacz boosted model \[\begin{equation} \hat{f}(x) = \sum_{b=1}^B\lambda\hat{f}^b(x) \end{equation}\]
Uczenie drzew klasyfikacyjnego metoda boosting przebiega w podobny sposób. Wynik uczenia drzew metodą boosting zależy od trzech parametrów:
- Liczby drzew \(B\). W przeciwieństwie do metody bagging i lasów losowych, zbyt duże \(B\) może doprowadzić do przeuczenia modelu. \(B\) ustala się najczęściej na podstawie walidacji krzyżowej.
- Parametru “kurczenia” (ang. shrinkage) \(\lambda\). Kontroluje on szybkość uczenia się kolejnych drzew. Typowe wartości \(\lambda\) to 0.01 lub 0.001. Bardzo małe \(\lambda\) może wymagać dobrania większego \(B\), aby zapewnić dobrą jakość predykcyjną modelu.
- Liczby podziałów w drzewach \(d\), która decyduje o złożoności drzewa. Bywa, że nawet \(d=1\) daje dobre rezultaty, ponieważ model wówczas uczy się powoli.
Implementację metody boosting można znaleźć w pakiecie gbm (Greenwell et al. 2019)
Przykład 5.3 Metodę boosting zastosujemy do zadania predykcji ceny mieszkań na przedmieściach Bostonu. Dobór parametrów modelu będzie arbitralny, więc niekoniecznie model będzie najlepiej dopasowany.
library(gbm)
boston.boost <- gbm(medv~., data = boston.train,
distribution = "gaussian",
n.trees = 5000,
interaction.depth = 2,
shrinkage = 0.01)
boston.boost## gbm(formula = medv ~ ., distribution = "gaussian", data = boston.train,
## n.trees = 5000, interaction.depth = 2, shrinkage = 0.01)
## A gradient boosted model with gaussian loss function.
## 5000 iterations were performed.
## There were 13 predictors of which 13 had non-zero influence.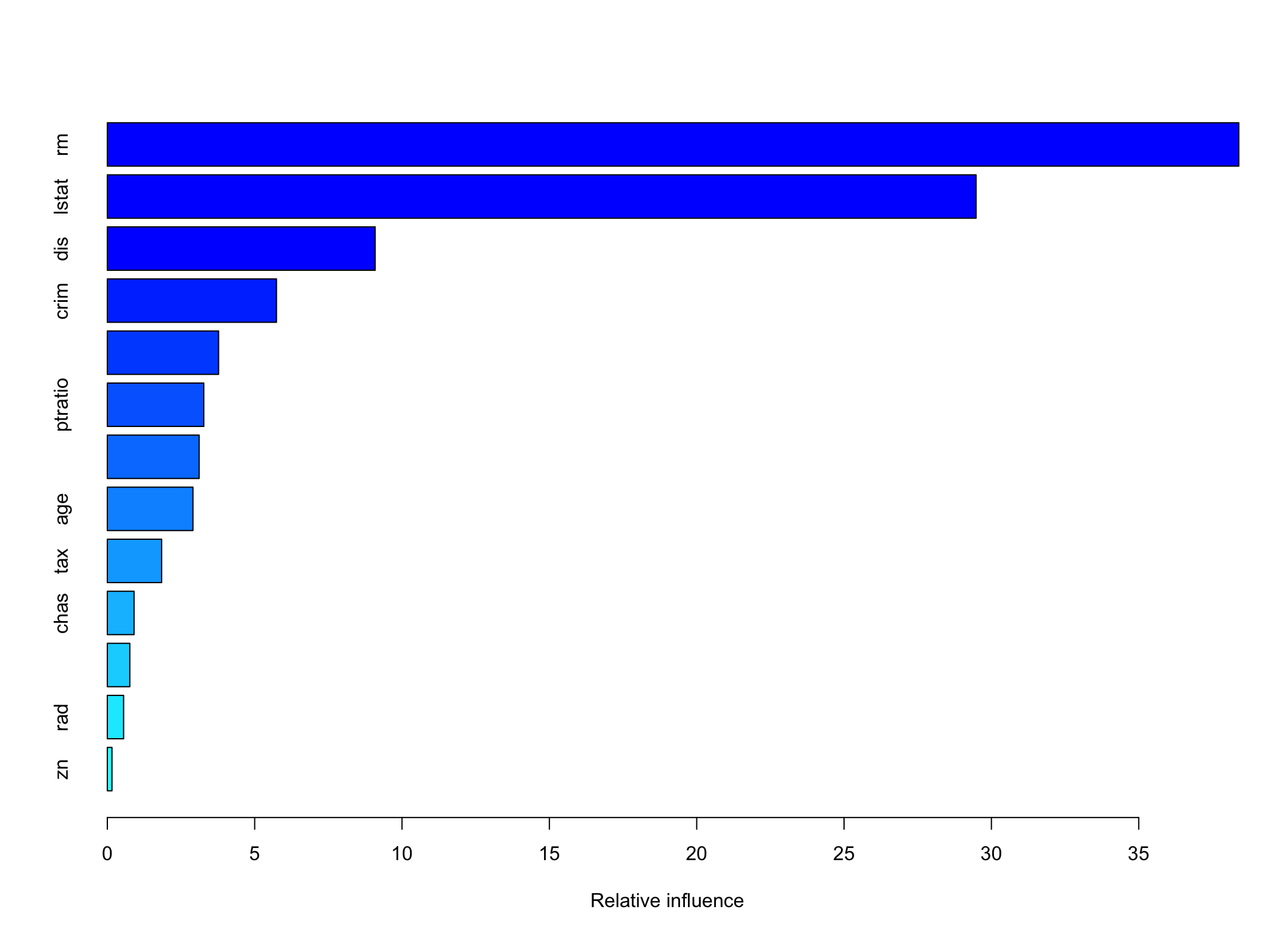
## var rel.inf
## rm rm 38.3955886
## lstat lstat 29.4805422
## dis dis 9.0886721
## crim crim 5.7399540
## nox nox 3.7754214
## ptratio ptratio 3.2740541
## black black 3.1164954
## age age 2.9063950
## tax tax 1.8433918
## chas chas 0.9067974
## indus indus 0.7627923
## rad rad 0.5523485
## zn zn 0.1575472Predykcja na podstawie metody boosting
boston.pred4 <- predict(boston.boost, newdata = boston.test, n.trees = 5000)
rmse(boston.pred4, boston.test$medv)## [1] 3.801233\(RMSE\) jest w tym przypadku nieco większe niż w lasach losowych ale sporo mniejsze niż w metodzie bagging. Wszystkie metody wzmacnianych drzew dają wyniki lepsze niż pojedyncze drzewa.
Bibliografia
chyba tylko dla drugiej metody istniej dobre polskie tłumaczenie nazwy - las losowy↩︎